O RFLP (Polimorfismo no Comprimento de Fragmentos de Restrição) é uma técnica da biologia molecular que analisa pedaços de DNA que foram cortados por enzimas de restrição (esses pedaços são chamados de fragmentos de restrição). O funcionamento é bem simples: existem enzimas que conseguem cortar o DNA em algumas regiões. Essas enzimas são chamadas de enzimas de restrição e apresentam vários tipos, que cortam apenas sequências reconhecidas por elas. Porém, o que fica entre essas regiões pode variar de tamanho. A primeira vez que pesquisadores sugeriram essa técnica foi em 1974 e em pouco tempo ela se tornou uma ferramenta eficaz para inúmeros procedimentos de biologia molecular e biotecnologia. Quatro anos depois da descoberta dessa técnica, os pesquisadores responsáveis (Arber, Nathans e Smith) receberam o prêmio Nobel em Medicina ou Fisiologia.
Para entendermos o funcionamento dessa técnica devemos entender o que é um marcador molecular: uma região do DNA será um marcador molecular quando ela apresentar características específicas para um grupo de análise (seja esse grupo uma espécie, uma família…). Assim, o RFLP será um marcador quando apresentar variabilidade suficiente que permita análises individuais ou populacionais.
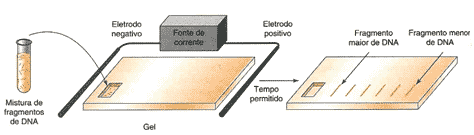
Ok, mas como verificar o tamanho desse fragmento de DNA? Bom, a resposta é simples. Existe uma técnica chamada Eletroforese em gel, que consiste na migração de uma molécula em um gel a partir da diferença de potencial elétrico. Para que isso ocorra é preparado um gel com agarose (polissacarídeo gelatinoso encontrado em algas) que prenderá as moléculas durante a migração. Após isso, é colocado o DNA em poços (pequenas cavidades no gel) e é aplicada uma corrente elétrica.
Fonte: http://www.sobiologia.com.br/conteudos/Biotecnologia/eletroforese.php
O DNA tem carga negativa, então ele será atraído pelo polo positivo. Entretanto, para chegar a esse polo, a molécula deve migrar através do gel. Os fragmentos de DNA menores irão migrar mais rapidamente do que os fragmentos maiores. Após terminar o tempo de “corrida”, é aplicada uma luz ultravioleta (UV) nesse gel, visto que antes da aplicação o DNA é tratado com uma molécula que “brilha” quando exposta a essa luz UV. Quando aplicada a luz, vê-se no gel algumas faixas de luz brilhantes, que correspondem ao DNA.
Mas como saber qual o tamanho exato desse DNA para que se possa prosseguir na análise? Para isso, utiliza-se um Ladder, que nada mais é do que uma sequência de DNA de tamanho conhecido, que aparecerá no gel. Assim, é possível comparar a amostra que o pesquisador desconhece com a conhecida, descobrindo assim o tamanho da amostra. Esse ladder é medido em pares de base, ou seja, quantas bases nitrogenadas (componentes do DNA) tem naquele fragmento.
Para facilitar ainda mais a visualização das bandas, faz-se uma técnica chamada de Southern Blot, que consiste em transferir o DNA do gel da eletroforese para uma membrana de nylon ou nitrocelulose. Cada indivíduo ou grupo apresentará um padrão de fragmentos (bandas), que é chamado de perfil de digestão, que é detectado pelo número e tamanho dos fragmentos gerados. Assim, essa técnica pode servir para identificar genótipos heterozigotos, visto que é um marcador codominante; para mapear o genoma de um grupo; para teste de paternidade; para detecção de doenças hereditárias e muitas outras aplicações.
Com isso, vemos que o RFLP é um bom marcador molecular, por apresentar diversidade em aplicações e ser um marcador codominante, ou seja, onde é possível se diferenciar homozigotos de heterozigotos. Este marcador pode apontar relações de cruzamento entre casais, determinar relações de parentesco e até auxiliar na genética forense para resolução de crimes. Porém, entre as desvantagens dessa metodologia podemos citar o fato desta ser cara para execução (demanda de aparelhos e reagentes caros), utilizar sondas que são tóxicas (necessitam de cuidado no manuseio) e é relativamente trabalhoso.
Texto por: Francisco Sassi